こんにちは。DryとWetで創薬を夢見るDrywettyです。今回は創薬アドベントカレンダーに参加したいと思い、Dryで遺伝子ネットワーク解析をテーマにしてみました。ネットワーク解析って何かすごいことがすぐにわかるわけではないんですが、インパクトのある図が作れるのが楽しくて好きなんです。この記事では、遺伝子ノックアウトした細胞で大きく影響を受けているサブネットワークを抽出する方法を紹介します。この解析ができると遺伝子ノックアウトだけではなく遺伝子過剰発現や化合物刺激をした際の解析にも応用ができます。
Cytoscapeに変動遺伝子をインポート
まずは変動遺伝子解析をして遺伝子にFDRやp-valueが計算されている表を準備します。サブネットワークの抽出にp-valueの値を使います。今回は例として2015年のCellにあったA Genome-wide CRISPR Screen in Primary Immune Cells to Dissect Regulatory Networksのsup. table 4の一つを使いました。今回はFDR (padj) < 0.05の94 genesに絞りました。
DE genes in sgRNA targeting OST subunits; 0h LPS
(上から5行を抜粋)
| gene | baseMean | log2FoldChange | lfcSE | stat | pvalue | padj |
| Manf | 279.0401 | 1.185907 | 0.110384 | 10.74348 | 6.36E-27 | 6.E-24 |
| Dnajc3 | 230.5844 | 0.923177 | 0.092108 | 10.02272 | 1.21E-23 | 7.E-21 |
| Creld2 | 178.4676 | 1.194198 | 0.109544 | 10.90154 | 1.13E-27 | 1.E-24 |
| Pdia6 | 661.7309 | 0.985884 | 0.088859 | 11.09498 | 1.33E-28 | 3.E-25 |
| Hspa5 | 1456.273 | 1.298899 | 0.125885 | 10.31817 | 5.83E-25 | 4.E-22 |
この表をgeneをノードとしてCytoscapeに取り込みます。Cytoscapeの基本的な使い方は統合TVを参照してください。
GeneMANIAのCytoscape Appで遺伝子間ネットワークを追加
遺伝子同士の関係性はCytoscape Appの一つであるGeneMANIAを使います。GeneMANIAはクエリした遺伝子同士に対してphysical interactionやco-expression、co-localizationなどの関係性を追加できます。GeneMANIA自体はwebベースのcytoscape.jsを用いたサイトが本家で遺伝子ネットワークの可視化をしてくれますが、今回はサブネットワーク抽出まで行いたいのでCytoscape Appで行います。
AppメニューからGeneMANIA>Local searchを選んで調べたい遺伝子群をコピペします。Local searchをする前に調べたい種のデータをダウンロードしておきましょう。ヒト、マウスそれぞれ2.5Gbくらいあります。
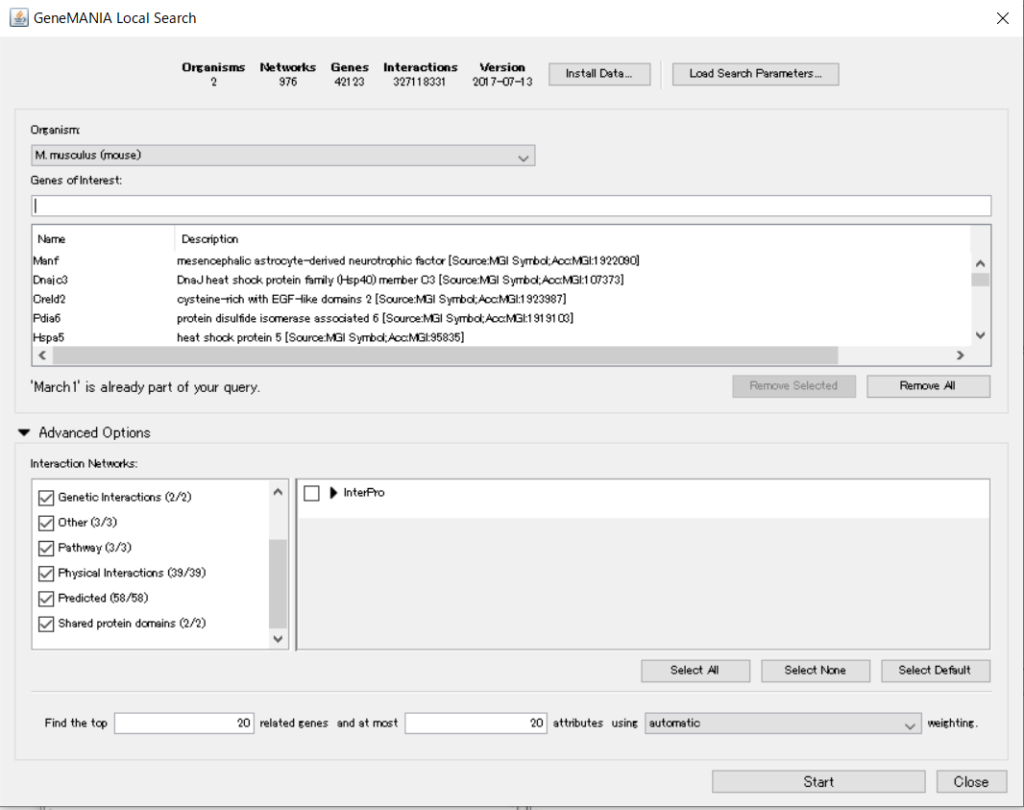
遺伝子群がインポートされたらinteractionを検索したいソースを選んで「Start」で検索します。Co-expressionやPredictedは検索でヒットするinteractionがかなり多くなってしまいますので今回は抜きました。
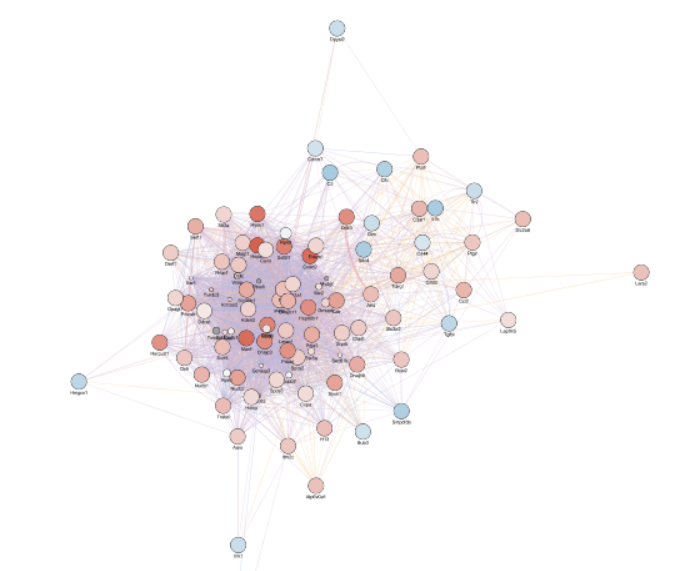
得られたネットワークは下図のとおり。いい感じの毛玉になりました。今回はlog2foldchangeで色付けしています。このネットワークから中でも変動が大きいサブネットワークをjActiveModulesで抽出します。
jActiveModules Cytoscape Appでサブネットワークの抽出
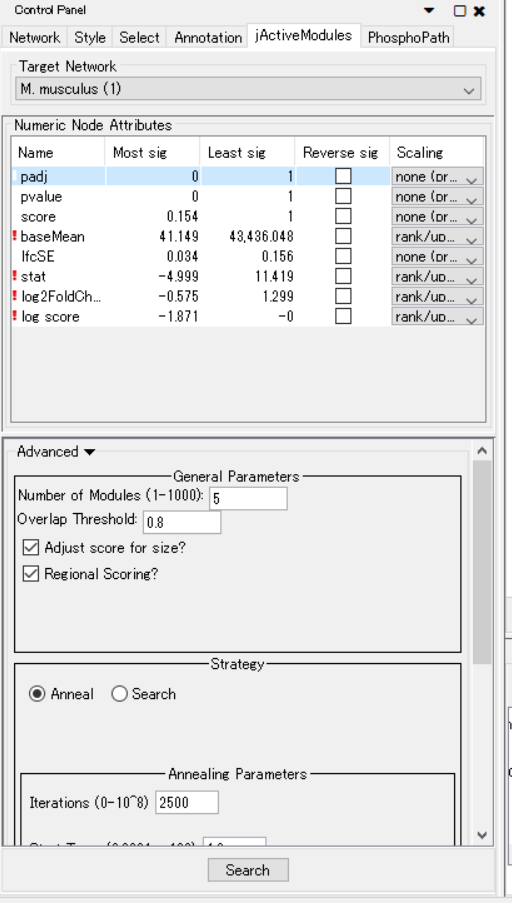
変動の大きいサブネットワークの抽出にはjActiveModulesというCytoscape Appを用います。jActiveModulesのアルゴリズム自体は2002年に発表されて焼きなまし法(simulated annealing)がベースの手法ですが、2017年のサブネットワーク抽出のベンチマーク論文でもよいスコアを出しています。
コントロールパネルのjActiveModulesタブを選択して、読み込んだネットワークを選択。active neworkの抽出に使いたいスコアを選択するのを忘れずに。クリックして青くなっているのが選択されている状態です。今回はpadjを選択しました。パラメーターについては今日は議論しません。Strategyがannealとsearchがあり、ここは大きく影響するので気を付けてください。ベンチマーク論文はanneal strategyがrecall rateが高くて高評価でしたが、遺伝子数の多いネットワーク(>5000 genes)ではマシンパワーが必要になると注意がありました。私のPCがしょぼいので今回はSearchで行きます。
結果、94 genes, 2829 interactionsだったネットワークから47 genes, 803 interactionsのサブネットワークが抽出されました。論文中に記載のあるHsp90b1やSec61a1、Ostcがみられるのである程度妥当な結果になったと思います。
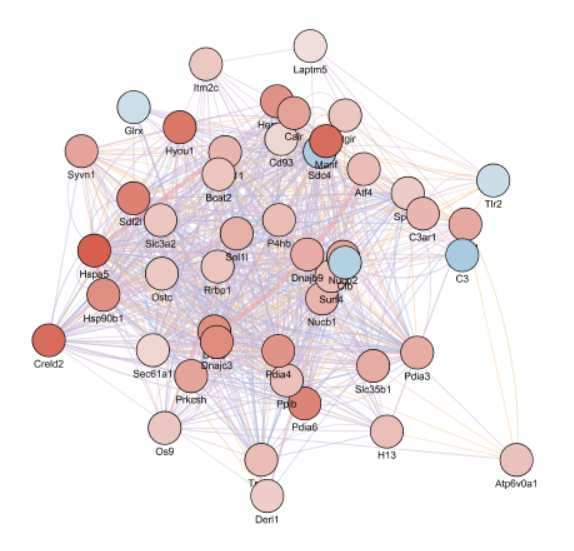
ここから本当にinteractionしている因子かどうかを調べるのは、 標的遺伝子に対する抗体を使って免疫沈降をしてMS解析をするなど、実験的な検証が必要になります。解析のその先が大事になるので解析系の文献を読んだらどう検証しているかは要注意です。検証までちゃんとしている論文は結構いいジャーナルに出ている傾向があります。読んでいただきありがとうございました!